Entwicklung eines Bewertungsverfahrens für Biodiversität und die Vitalität von Bestäubern in Agrarflächen
Rund 70 % der weltweit angebauten Kulturpflanzen profitieren von Bestäubern. Jedoch sind Bienen wie auch andere Tiere durch landwirtschaftliche Stressoren gefährdet. Detailliertes Wissen über das Sammelverhalten in verschiedenen Landschaftsszenarien ist also notwendig, um zu gewährleisten, dass die Bestäubung durch Bienen als wichtige Ökosystemdienstleistung auch in Zukunft bereitgestellt werden kann. Dafür geeignete Sequenzierungsmethoden durchzuführen und die entstandenen Sequenzdaten so aufzubereiten, dass diese Methoden nicht nur evaluiert werden können, sondern auch auswertbare Ergebnisse entstehen, ist ein wichtiger Schritt zur Etablierung eines nachhaltigen Naturschutzes. Längerfristig soll die Nanoporensequenzierung vor allem für Sofort-Untersuchungen im Feld eingesetzt werden. Das heißt, dass im Feld die Proben entnommen, aufbereitet und mit dem MinION sequenziert werden, um schnell Ergebnisse zu erhalten und entsprechend zu handeln können. Bereits 2015 wurde eine bahnbrechende diagnostische Untersuchung im westafrikanischen Ebolagebiet Guinea durchgeführt, indem in Echtzeit mit dem MinION das Ebolavirus sequenziert wurde. Demzufolge konnten schnelle Maßnahmen zur Versorgung der Patienten getroffen werden1.
Unser Beitrag
Biomonitoring anhand von Umwelt-DNA-Analysen durchzuführen, zeichnet sich durch eine besonders tier- und umweltfreundliche Arbeitsweise aus2. Dabei wird auf DNA-Material zurückgegriffen, dass sich in der Umwelt befindet wie zum Beispiel in Bodenproben, Flusswasser oder Sediment. In Pollen, der an Bestäubern haftet, befindet sich ebenfalls DNA3. Auf den Sammelflügen von Bienen etwa bilden sich sogenannte Pollenhöschen an den Beinen der Tiere, die für die Versorgung der Brut in den Nestern bzw. Stöcken eingelagert werden4. Eine genetische Analyse der gesammelten Pollen-DNA lässt Rückschlüsse auf das Sammelverhalten zu. Je nach Positionierung der Bienenstöcke können so ökologische Fragestellungen bearbeitet werden.
Mit der Etablierung genomischer Untersuchungen lassen sich genauere und aufschlussreichere Aussagen über biologische Fragestellungen treffen2. So können Ökologen bzw. Agrarwissenschaftler ableiten, welche Sammelstrategie Bestäuber in Anbetracht der geografischen Lage ihrer Bienenstöcke (massenblühende Felder, Weiden, Obstbaumplantagen usw.) verfolgen. So erfordert diese Art des Biomonitoring keinen störenden oder gar zerstörenden Observationsaufwand im Feld.
Bisherige Ergebnisse
 Ausschnitte der Erbinformation (DNA) des von Hummeln und Honigbienen gesammelten Pollen wurden bereits mittels zweier DNA-Sequenziermethoden in Zusammenarbeit mit dem Lehrstuhl für funktionelle Agrarökologie der Georg-August-Universität Göttingen untersucht. Neben den Resultaten einer gängigen Polymerase-basierten Sequenzierungsmethode haben wir die neuartige Nanoporensequenzierung genutzt, um Pollen-DNA zu untersuchen5. Anhand eines speziellen Abschnittes im Pollen-Erbgut, der sogenannten ITS2-Region, wurden die aufgesuchten Pflanzen bioinformatisch identifiziert und die Pollenanteile quantifiziert. Dabei ergab sich, dass bei gleichen Standortbedingungen Hummeln ein breiteres Spektrum an Pflanzen besuchten als Honigbienen. Zudem beschränken sich beide nicht auf massenblühende Feldfrüchte wie Raps – immer werden, trotz Mehraufwand, weitere Blühpflanzen besucht. Dies kann als weiteres Argument für die Etablierung von Ackerrandstreifen angeführt werden.
Ausschnitte der Erbinformation (DNA) des von Hummeln und Honigbienen gesammelten Pollen wurden bereits mittels zweier DNA-Sequenziermethoden in Zusammenarbeit mit dem Lehrstuhl für funktionelle Agrarökologie der Georg-August-Universität Göttingen untersucht. Neben den Resultaten einer gängigen Polymerase-basierten Sequenzierungsmethode haben wir die neuartige Nanoporensequenzierung genutzt, um Pollen-DNA zu untersuchen5. Anhand eines speziellen Abschnittes im Pollen-Erbgut, der sogenannten ITS2-Region, wurden die aufgesuchten Pflanzen bioinformatisch identifiziert und die Pollenanteile quantifiziert. Dabei ergab sich, dass bei gleichen Standortbedingungen Hummeln ein breiteres Spektrum an Pflanzen besuchten als Honigbienen. Zudem beschränken sich beide nicht auf massenblühende Feldfrüchte wie Raps – immer werden, trotz Mehraufwand, weitere Blühpflanzen besucht. Dies kann als weiteres Argument für die Etablierung von Ackerrandstreifen angeführt werden.
Durch bioinformatische Analysen der Sequenzdatensätze wurde erkannt, dass die moderne Nanoporen-Sequenzierung für die Untersuchung gleichwertig geeignet ist, aber Vorteile durch die Portabilität sowie den geringeren Zeit- und Kostenaufwand aufweist6.
1 Quick, J.; Loman, N. & Duraffour, S. et al. (2016): Real-time, portable genome sequencing for Ebola surveillance. Nature. 530(7589):228-232. doi:10.1038/nature16996
2 Keller, A.; Grimmer, G. & Sickel, W. et al. (2016) DNA-Metabarcoding-ein neuer Blick auf organismische Diversität. BioSpektrum. 22: 147. DOI: 10.1007/s12268-016-0669-0
3 Taberlet, P.; Bonin, A. & Zinger, L. et al. (2018): Environmental DNA: For Biodiversity Research and Monitoring. Oxford University Press
4 Jarczok, R. (Rdk.) (2018) Arbeiten mit Bienen – Imkern. Garant Verlag GmbH
5 Leidenfrost, R.M.; Bänsch, S.; Prudnikow, L. et al. (2020) Analyzing the Dietary Diary of Bumble Bee. Front Plant Sci 11:1572. DOI: 10.3389/fpls.2020.00287
6 van Dijk, E. L.; Jaszyszyn, Yan; Naquin, Delphine et al. (2018): The third Revolution in Sequencing Technology. Trends in Genetics. 34(9):666-681. doi: 10.1016/j.tig.2018.05.008
Im Mittweidaer Team forschen Lisa Prudnikow, Robert Leidenfrost und Professor Röbbe Wünschiers. Das Forschungsprojekt wird vom Sächsisches Staatsministerium für Wissenschaft, Kultur und Tourismus und dem Europäischen Sozialfond unterstützt.
Aktuelles

Methodenoptimierung der Isolierung von DNA zur Mikrobiomanalyse von Wildbienen-Brutzellen und Individuen
Forscher Nachwuchs | NWK 2025 | Vanessa Bergler untersucht die Mikroflora der Wildbienen
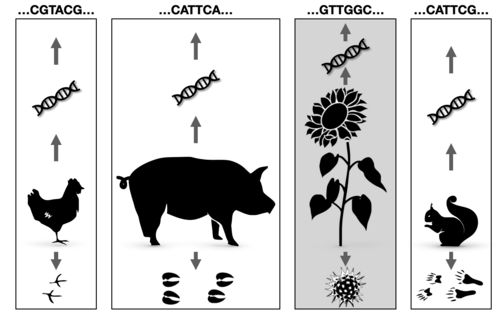
Metabarcoding zur Identifikation von Arten
In ihrer aktuellen Veröffentlichung mit dem Titel A primer on pollen assignment by nanopore-based DNA sequencing im Journal Frontiers in Ecology and Evolution zeigen Lisa Prudnikow, Birgit Pannicke und Professor Röbbe Wünschiers, wie mit der DNA aus...

1. Fachtag der Bienengesundheit an der Hochschule Mittweida
Bienenfreunde aller Art kommen zur Hochschule Mittweida, um über Bienengesundheit zu diskutieren.

„Summ, summ, summ – Bienchen summ herum“
1. Fachtag der Bienengesundheit an der Hochschule Mittweida am 19. November 2022 nimmt die fleißigen Insekten unter die Lupe

Beurteilung des Einflusses von Agrarumweltmaßnahmen auf Honigbienen basierend auf genetischer Pollenanalyse mit Nanoporensequenzierung
Forscher Nachwuchs | Birgit Pannicke nimmt Honigbienen-Pollenhöschen unter die Lupe

Volkszählung bei Honigbienen – Gemeinsam gegen Varroamilben
Für das Bienensterben gibt es vielfältige Gründe: veränderte Landnutzung, intensivierte Landwirtschaft, Pandemien. Die Milbe Varroa destructor gehört zu letzteren – und der Name ist Programm. Das rund 1,5 Millimeter große Weibchen lebt parasitär in...

SAVE THE DATE: Varroamilben-Zähltage bei Honigbienen

Göttinger Pollen im Mittweidaer Labor
Die Masteraspirantinnen Birgit Pannicke und Lisa Prudnikow aus der Forschungsgruppe um Professor Röbbe Wünschiers beschäftigen sich mit der genetischen Pollenanalyse.

Die Speisekarte von Hummeln
Bei Bestäubung denken die meisten an Bienen. Hummeln (engl. bumble bee) sind aufgrund ihrer Ernährungsgewohnheiten mindestens ebenso wichtige Bestäuber und spielen als solche eine bedeutende Rolle für Ökosysteme. Und der Pollen stellt eine wichtige...

20. Nachwuchswissenschaftlerkonferenz
Die Nachwuchswissen- schaftlerkonferenz (NWK) feierte in diesem Jahr ihr 20. Jubiläum an der Hochschule Merseburg. Für zwei Tage trafen sich Wissenschaftler aus verschiedenen Fachgebieten, um ihre aktuellen Forschungsgebiete vorzustellen und der...
Publikationen
Leidenfrost RM, Bänsch S, Prudnikow L, Brenig B, Westphal C and Wünschiers R (2020) Analyzing the Dietary Diary of Bumble Bee. Front. Plant Sci. 11:287. doi: 10.3389/fpls.2020.00287
Konferenz- und Tagungsbeiträge
Development of a genetic monitoring test for the investigation of pollinator-plant interaction
Lisa Prudnikow, Röbbe Wünschiers & Catrin Westphal
26. Interdisziplinäre Wissenschaftliche Konferenz Mittweida, Hochschule Mittweida/Germany, digital; 14th and 15th of April 2021
Genetic Pollen Analysis
Lisa Prudnikow
Züchtertagung der Gemeinschaft der europäischen Buckfastimker e.V., digital; 6th of March 2021
Pollen-DNA-Sequenzierung zur Analyse des Sammelverhaltens von Honigbienen und Hummeln
Lisa Prudnikow, Robert Leidenfrost & Röbbe Wünschiers
5. Konferenz für studentische Forschung, Universität Hohenheim/Germany, digital; 17-18th of September 2020
Leibniz Institute of Plant Genetics and Crop Plant Research, Gatersleben/Germany; 18-22nd of March 2019
Herkunftsbestimmung von Pollen zur Analyse des Sammelverhaltens von Honigbienen und Hummeln durch Nanoporen- und Polymerase-basierte Sequenzierungsverfahren
Prudnikow, Lisa; Leidenfrost, Robert; Wünschiers, Röbbe
21ste Nachwuchswissenschaftlerkonferenz NWK20, Jena/Germany; Accepted 20.03.2020
Herkunftsbestimmung von Pollen zur Analyse des Sammelverhaltens von Honigbienen und Hummeln.
Lisa Prudnikow, Robert Leidenfrost & Röbbe Wünschiers
20ste Nachwuchswissenschaftlerkonferenz NWK19, Merseburg/Germany; 18-19th of June 2019
Reading Plant ITS2-Sequences with Nanopores and Polymerases: First Insights to Pros and Cons.
R Leidenfrost, S Bänsch, L Prudnikow, C Westphal & R Wünschiers
15th Gatersleben Research Conference on Applied Bioinformatics in Crops
Leibniz Institute of Plant Genetics and Crop Plant Research, Gatersleben/Germany; 18-22nd of March 2019


