Metabarcoding zur Identifikation von Arten
Metabarcoding zur Identifikation von Arten
In ihrer aktuellen Veröffentlichung mit dem Titel A primer on pollen assignment by nanopore-based DNA sequencing im Journal Frontiers in Ecology and Evolution zeigen Lisa Prudnikow, Birgit Pannicke und Professor Röbbe Wünschiers, wie mit der DNA aus Pollen die Pflanzenquelle des Pollens detektiert werden kann.
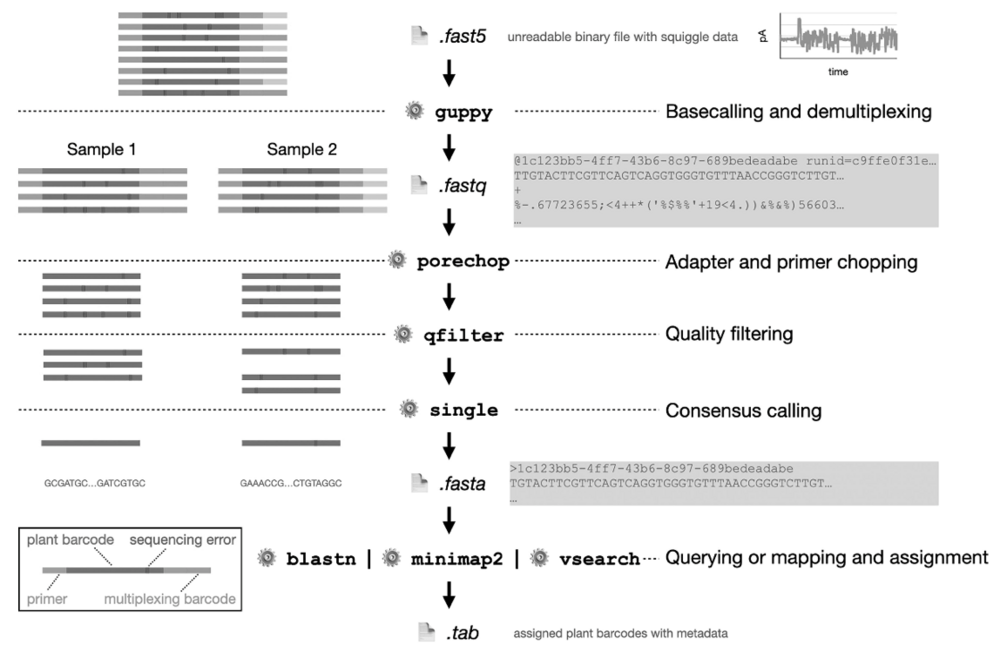
Der Fokus liegt dabei auf der Sequenzierung der DNA mit der Nanoporentechnologie. Diese Methode ist relativ billig und einfach in der Anwendung und führt zur Zeit zu einer "Demokratisierung" der DNA-Analyse. Verglichen mit anderen, schon länger etablierten DNA-Sequenziertechnologien ist die Nanoporen-basierte DNA-Sequenzierung aber ungenauer. Von der Mehrheit der Wissenschaftler wird deshalb bezweifelt, dass sie sich beispielsweise für das Metabarcoding zur Identifikation von Arten eignet.
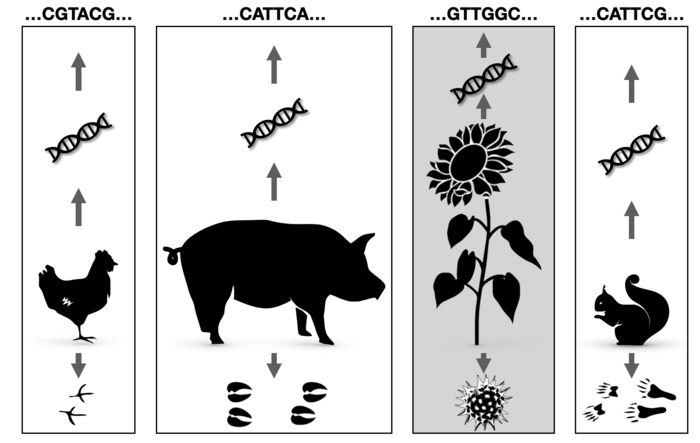
Beim Barcoding wird beispielsweise aus Fragmenten von Insekten, Pilzen oder Bakterien die Erbsubstanz DNA extrahiert und mit der DNA-Sequenzierung ein bestimmter Abschnitt auf der DNA, der Barcode, "gelesen". Die gelesene DNA-Sequenz wird dann mit Datenbanken abgeglichen und so die Quelle der DNA identifiziert – wenn der entsprechende Barcode bereits in der Datenbank enthalten ist. Das Metabarcoding erweitert diese Technologie auf gemischte Proben.
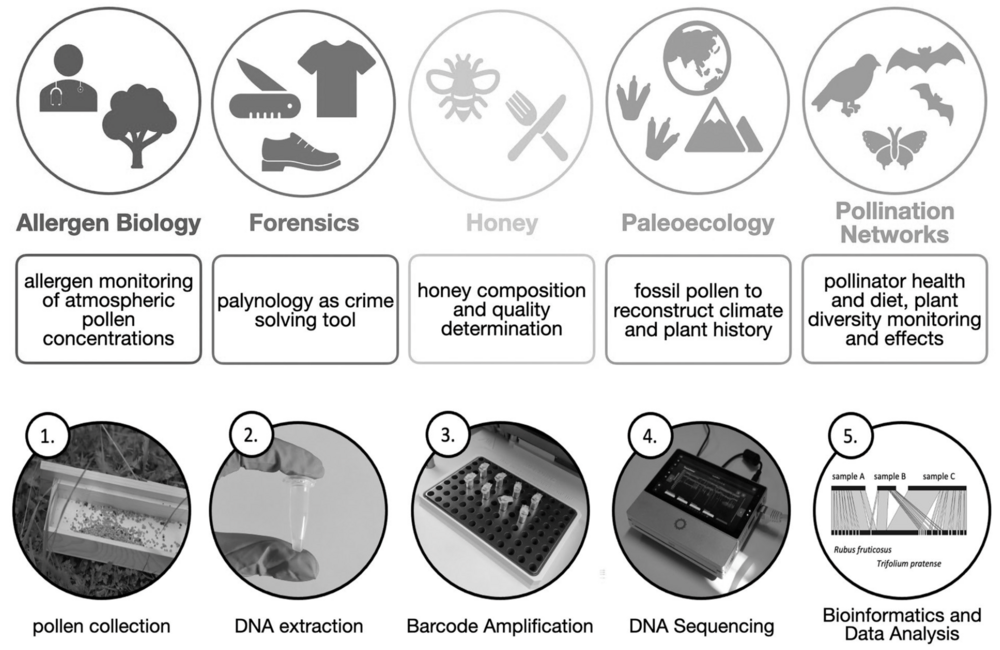
In der Forschungsgruppe um Professor Wünschiers ist die Methode des Metabarcoding seit mehreren Jahren bei der Zuordnung von Pollen zu Pflanzen im Einsatz. Der Pollen stammt dabei zumeist von Bienen und dient der Beantwortung ökologischer Fragestellungen.
In der Anfangsphase standen vor allem Vergleichsstudien der Nanoporen-Sequenzierung mit etablierten Verfahren im Fokus. Dabei zeigte sich, dass die schlechtere Qualität der Nanoporen-Sequenzdaten durch eine gezielte bioinformatische Aufarbeitung der Daten kompensiert werden kann. Dann liefert die die günstige und mobil einsetzbare Technik vergleichbare Ergebnisse wie etablierte Verfahren. Die Arbeiten der AG Wünschiers tragen dazu bei, Möglichkeiten und Grenzen der neuen Sequenziertechnologie aufzuzeigen. Vor allem machen sie deutlich, dass sowohl Proben- als auch Methodenkenntnis notwendig sind, um optimale Ergebnisse zu erzielen. Die Nanoporen-DNA-Sequenzierung ist in dieser Hinsicht besonders "Fehleranfällig" – frei nach dem Motto: Garbage in, garbage out.
Mit der aktuellen Publikation zeigen Lisa Prudnikow, Birgit Pannicke und Professor Röbbe Wünschiers, was es bei der Probenvorbereitung und Datenanalyse zu beachten gilt. Natürlich lassen sich die Erfahrungen und Erkenntnisse auch auf anderes Probenmaterial anstelle von Pollen übertragen. So spielt das Metabarcoding gerade im Kontext der Biodiversitätsforschung eine wichtige Rolle.
Prudnikow, Pannicke & Wünschiers (2023) A primer on pollen assignment by nanopore-based DNA sequencing. Frontiers in Ecology and Evolution, Vol. 11. doi: 10.3389/fevo.2023.1112929