Strategien zur genetischen Klassifikation des Hasenpesterregers
Strategien zur genetischen Klassifikation des Hasenpesterregers
Die Hasenpest (Tularämie) ist eine vom Bakterium Francisella tularensis verursachte Infektionskrankheit, die bei freilebenden Nagern und Hasenartigen häufig tödlich verläuft.
Sie ist eine Zoonose, d.h. sie kann auch den Menschen betreffen. Beim Menschen verläuft die Krankheit schwer und, insbesondere unbehandelt, häufig lebensbedrohlich. Zudem ist der Nachweis des Erregers nach dem deutschen Infektionsschutzgesetz meldepflichtig. Dies liegt darin begründet, dass die Bakterien hoch infektiös sind und daher als Biowaffe eingesetzt werden könnten. Eine schnelle und aussagekräftige Diagnostik ist entsprechend erforderlich. Dabei geht es auch darum, ob ein in Deutschland unbekannter Subtyp die Erkrankung verursacht.
Zuständig für die Klassifikation des verantwortlichen Subtyps ist das Friedrich-Loeffler-Institut in Jena. Die aktuelle Diagnostik beginnt mit der Anzucht der Bakterien in einem Hochsicherheitslabor in Jena. Anschließend werden dort Proteine der Bakterienkultur isoliert und mittels Massenspektroskopie eine Art „Fingerabdruck“ erzeugt. Das Prozedere dauert mehrere Tage.
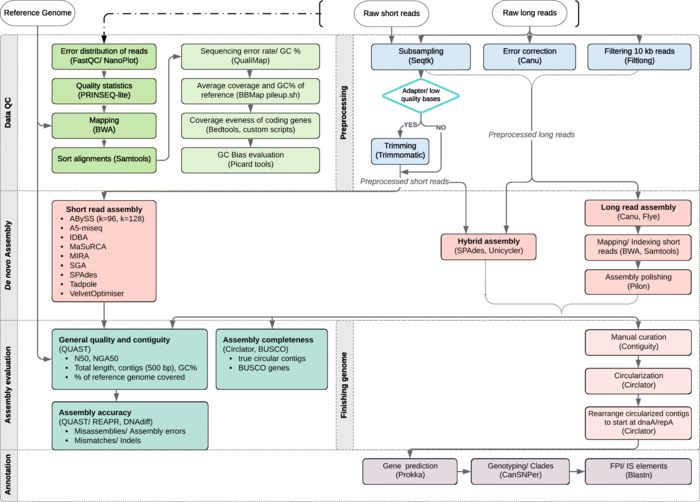
Seit einigen Jahren evaluiert die Forschungsgruppe um Professor Wünschiers, ob das Verfahren durch einen DNA-Test beschleunigt werden kann. Im Zentrum eines Kooperationsprojektes mit dem Friedrich-Loeffler-Institut in Jena (Bundesforschungsinstitut für Tiergesundheit) und dem Bundesinstitut für Risikobewertung in Berlin steht daher die Genomanalyse. Die Etablierung der Nanoporen-basierten DNA-Sequenzierung in Mittweida durch den Doktoranden Robert Leidenfrost ermöglicht es, hier einen wichtigen Beitrag leisten. Die Ergebnisse der umfangreichen Studie wurden im November 2021 im Fachmagazin BMC Genomics der Öffentlichkeit zugänglich gemacht. Dabei wurden fünf verschiedene Techniken der DNA-Sequenzierung und zahlreiche bioinformatische Methoden verglichen. Wir haben insbesondere untersucht, ob die genetische Struktur des sogenannten Francisella Pathogenicity Island (FPI) korrekt aufgeklärt wurde. Das FPI enthält für die Pathogenität verantwortliche Gene. Dabei zeigte sich, dass die Kombination von zwei Sequenziertechniken die besten Ergebnisse lieferte.
Das Forschungsprojekt wurde vom Sächsisches Staatsministerium für Wissenschaft, Kultur und Tourismus und dem Europäischen Sozialfond unterstützt.
Neubert K, Zuchantke E, Leidenfrost RM, Wünschiers R, Grützke J, Malorny B, Brendebach H, Al Dahouk S, Homeier T, Hotzel H, Reinert K, Tomaso H, Busch A. (2021) Testing assembly strategies of Francisella tularensis genomes to infer an evolutionary conservation analysis of genomic structures. BMC Genomics. 14;22(1):822. doi: https://doi.org/10.1186/s12864-021-08115-x.