Datenmassen richtig fassen für Biowissenschaftler
Es gibt Bioinformatiker, die entwickeln bioinformatische Methoden. Der viel größere Teil der Lebenswissenschaftler, etwa Biotechnologen oder Biologen, nutzt dagegen bioinformatische Methoden. Das die digitale Datenverarbeitung auch für Biotechnologen immer wichtiger wird, das lernen unsere Studierenden in ihrem Studium.
Professor Röbbe Wünschiers ist Biologe und in seiner Forschung ist die Anwendung bioinformatischer Methoden so wichtig wie der Umgang mit der Pipette. Doch während die Handhabung einer Pipette recht schnell erlernt ist, liegen bioinformatische Methoden nicht so leicht in der Hand. Man muss ein Basiswissen im Umgang mit Daten am Computer haben. Und Daten werden in den Lebenswissenschaften in riesigen Mengen erzeugt.
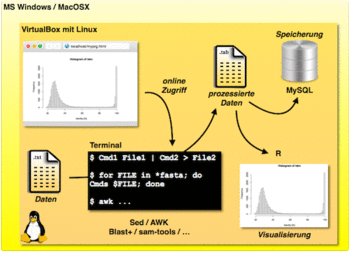
Vom Umgang mit Daten und der Anwendung bioinformatischer Methoden auf biologische Datensätze handelt das Lehrbuch "Wiley Schnellkurs Bioinformatik für Anwender" von Professor Röbbe Wünschiers. Er verarbeitet darin seine Erfahrungen aus 20 Jahren Forschung und Lehre. Der Untertitel "Datenmassen richtig fassen" beschreibt das Ziel des Buches sehr gut. Röbbe Wünschiers erklärt darin, wie Sequenz-, Struktur- und anderen Daten unter Linux prozessiert werden können. Linux, wie auch Apple's MacOSX, basieren auf dem Unix Betriebssystem und sind die beliebteste Entwicklungsplatform für Bioinformatiker. Linux, ebenso wie alle im Buch vorgestellte Software, sind kostenlos. Daher beginnt der praktische Teil des Buches mit einer Anleitung, wie man unter Windows oder MacOSX Linux als virtuelle Maschine installiert und wie Linuxtools wie Sed oder die einfache Programmiersprache AWK bei der Datenanalyse helfen können. Weitere Themen sind das Datenbanksystem MariaDB/MySQL, die Programmierumgebung R für statistisches Rechnen und Datenvisualisierung, die Textsatzsprache LaTeX und das Einrichten eines eigenen Webservers.
Der Hauptteil des Buches zeigt exemplarisch die Anwendung des Erlernten an fünf biologischen Beispielen aus der forensischen Mikrobiologie und Sequenzdatenanalyse. So wird etwa den Fragen nachgegangen, worin sich der Erreger der Spanischen Grippe vom heutigen Grippevirus unterscheiden, wie die Stoffwechselvorgänge in einer Biogasanlage analysiert und auf
einer Stoffwechselkarte darstellt werden können oder wie man Einzelnukleotidvarianten (SNPs) mittels der Sequenzierung des Ebolagenoms analysieren kann.
Alle notwendigen Dateien, sowie alle Abbildungen des Buches und weitere Informationen finden sich auf der Webseite datenmassen.de